Med:一次识别18种特定疾病/病原体!多RNA分子特征检测开辟儿童发热性疾病诊断新途径
时间:2023-09-07 22:55:21 热度:37.1℃ 作者:网络
近期,流感、手足口、疱疹性咽峡炎、支原体肺炎等多种传染病高发,由于强传染性,经常出现呈现出聚集性感染,一个孩子生病了,可能一个幼儿园、一个班都要面临停课。
儿童的免疫功能和代谢水平和成年人有较大区别,更容易感染一些在成年人群中不容易引起注意的病原体。通常儿童会被一些传染病和炎症性疾病所影响,表现出来最常见的症状为发烧,但是临床医生很难通过发热来可靠区分病毒感染、细菌感染或是炎症性疾病。
由于现有诊断方法的局限性,只有不到50%因发烧而前往急诊室的儿童能够得到明确的最终诊断。鉴于诊断的不确定性,许多没有细菌感染的患者不必要地接受广谱抗生素治疗,以降低漏诊严重细菌感染的风险,从而导致日益严重的抗菌药物耐药性问题。另一方面,由于诊断工作的不足,导致部分儿童因病情延误。如何识别并妥善治疗少数严重细菌感染和炎症性疾病患者,同时避免过度治疗大多数患有自限性疾病(通常是病毒性疾病)的患者,对临床来说是一个相当大的挑战。
基因表达芯片和RNA测序(RNA-seq)提供了一种潜在的替代诊断方法,通过检测感染性或炎性疾病患者血液中宿主基因表达的独特模式进行诊断,绕过了直接病原体检测。研究人员假设,通过在患者血液中检测有限数量的基因转录物,可以同时区分多种感染性和炎性疾病。

近期,由英国伦敦帝国理工学院研究人员领导的一个国际研究团队开发并验证了一种诊断方法,能够同时检测和区分18种传染性或炎症性疾病,包括B族链球菌(GBS)、呼吸道合胞病毒(RSV)和结核病。该研究结果发表在Cell旗下期刊Med上,题目为“Diagnosis of childhood febrile illness using a multi-class blood RNA molecular signature”。研究人员基于一种多类监督机器学习方法鉴定了161个转录本,可将患者分为18个疾病类别,反映了患者的致病病原体和特定疾病,以及对包括细菌感染、病毒感染、疟疾、结核病或炎症性疾病在内的广泛可靠预测。
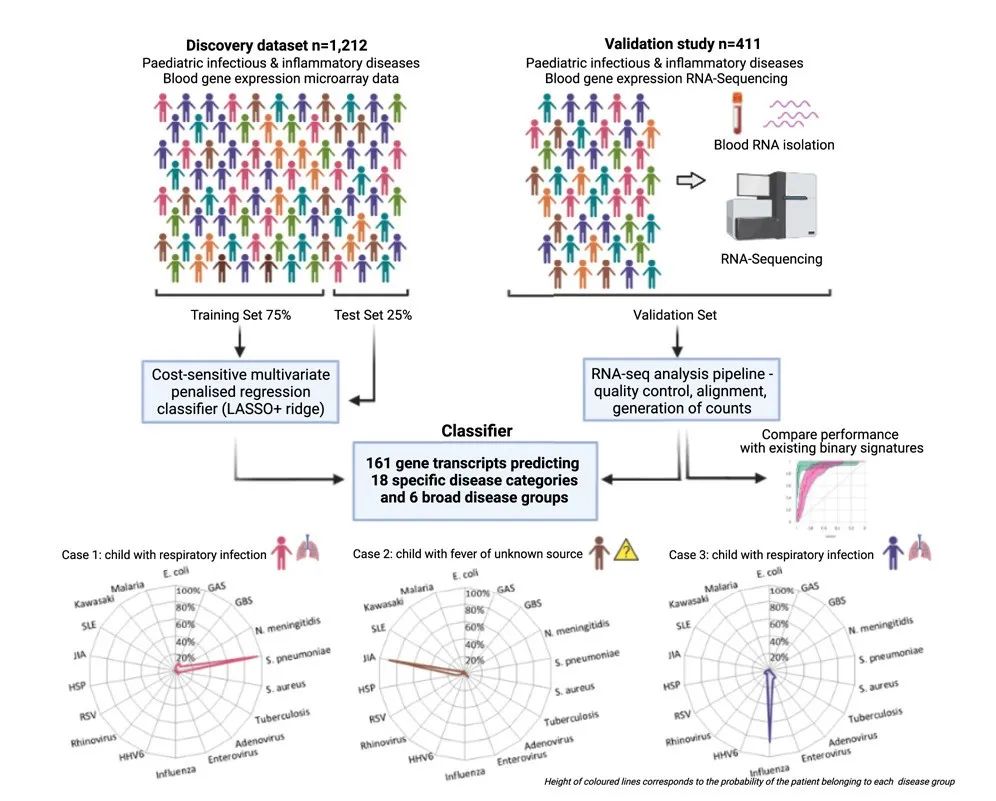
图1. 使用血液进行儿童疾病诊断的概要
建立儿童传染病多RNA特征诊断方法
为了探索利用有限数的RNA转录本对发热性疾病进行分类的可行性,研究人员基于一种多类监督机器学习方法合并分析了公开可用的芯片数据集。具体而言,是由12个公开可用的基因表达芯片数据集(n=1212)组成的数据集用于发现生物标志物组(图1),这些数据被随机分成占比75%和25%的子集,分别使用分层保留来维持类别比例进行训练和测试(图1)。
在发现数据集中,研究团队通过重复的交叉验证选择最佳方法来获得发热性疾病的多类特征。在比较的五种多元惩罚回归(penalized regression)方法中,LASSO + Ridge得到的模型具有良好的分类性能,同时具有成本效益。随后,研究人员在75%的发现集中应用多项LASSO+Ridge惩罚回归来识别由161个探针组成的RNA转录本panel,用于区分18种疾病类别(图1)。25%测试集的预测概率用于导出混淆矩阵,通过采用预测概率最高的类别来对每个样本进行离散类别预测(图2)。
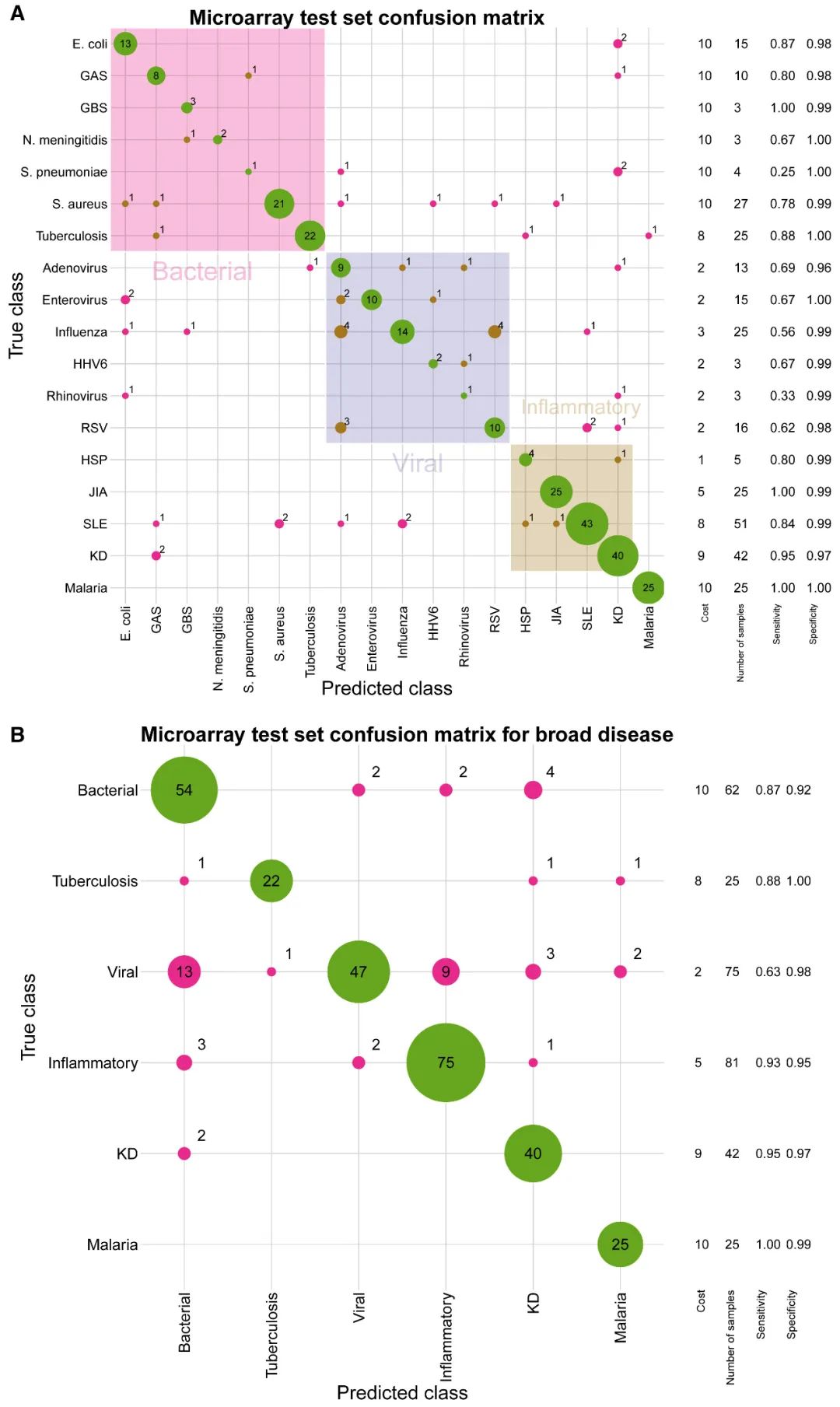
图2. 基因表达芯片检测预测的混淆矩阵
预测具有临床意义的广泛疾病类别
研究团队分析了上述生物标志物panel是否也可以用于广泛疾病类别的可靠预测。使用多项Ridge回归重新调整161个转录本的系数,使panel能够预测广泛的疾病类别:炎症性疾病、病毒感染、细菌感染、川崎病、疟疾和结核病。在考虑每个患者最可能的疾病类别和单独怀疑类别时,上述构建的模型准确地预测了这六种疾病类别。这些预测使模型能够反映临床决策中使用的诊断分类,同时解决多个临床问题,为临床团队提供每个患者疾病类别的概率。
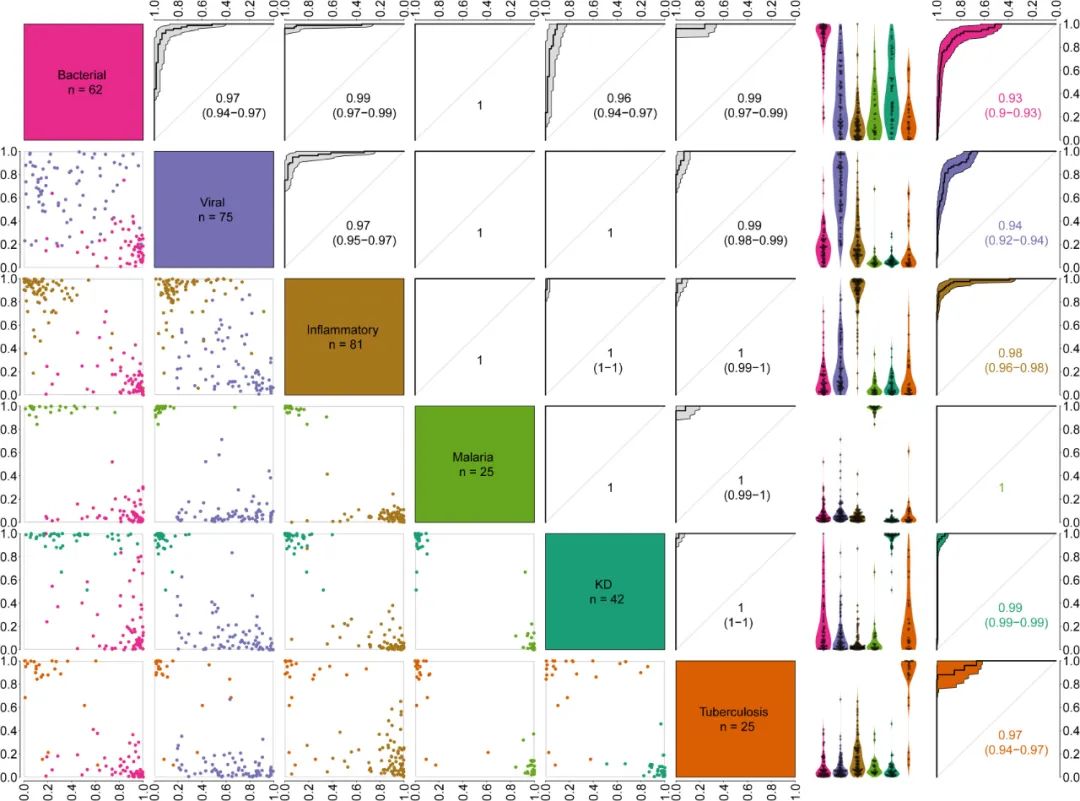
图3.芯片检测对广泛的疾病类别进行预测
使用RNA-seq的独立研究验证预测性能
另一方面,研究人员使用RNA定量平台评估了新RNA特征诊断方法在独立患者队列中的预测性能。研究人员使用了新生成的全血RNA-seq数据集,包括411名患有一系列传染病或炎症性疾病的儿科发热患者,涵盖发现数据集中所有六种广泛的诊断类别以及18种特定诊断类别中的13种(图1)。
结果显示,161个芯片探针被唯一地定位到155个基因,其中10个基因在RNA-seq数据集中没有足够的reads数来进行可靠的定量,最终在RNA-seq数据集中确认145个基因,支持了该方法的临床有效性、稳健性和可重复性。(图3、图4)
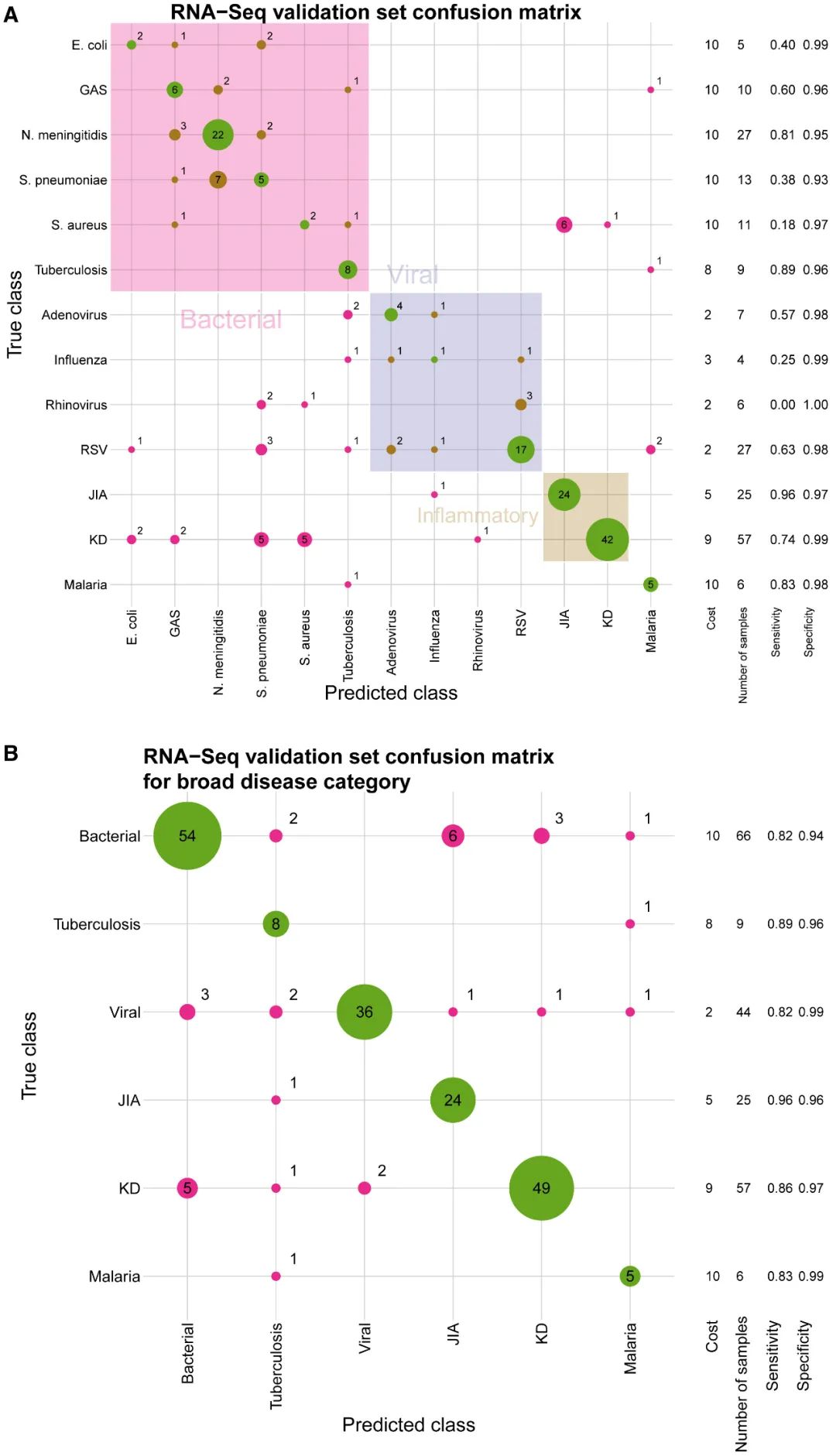
图3.验证队列中145-转录本的表现

图4.广泛疾病类别的RNA-seq验证集预测
该论文的共同高级作者、帝国理工学院传染病系高级讲师Myrsini Kaforou 博士表示:“这项工作使我们能够根据161个基因识别多种疾病的分子特征,其中包括人类基因组中的数千个基因。通过在同一次检测中同时区分多种疾病,我们开发了一个更全面、更准确的模型,与临床医生的诊断方式相一致。”
与已发布传染病特征的比较
此外,研究人员将该RNA特征诊断与已发表的一对多特征诊断进行了对比。使用相同的50%的RNA-seq数据集重新拟合每个线性模型的系数,并使用剩余50%的受试者工作特征(ROC)曲线评估性能。Wright签名和多类签名的Kawasaki分量之间的ROC曲线下面积(AUC)没有显著差异。对于结核病,多类生物标志物组在AUC方面表现优于单类Sweeney特征,但与Anderson特征的AUC没有显著差异。新方法在识别病毒感染和细菌感染方面具有显著性,但在区分细菌感染和病毒感染方面没有显著性。这反映了在该研究中纳入额外的疾病组对直接进行细菌-病毒AUC比较只有很小的影响,但使用成本敏感方法可以提高对细菌感染的敏感性。(图5)
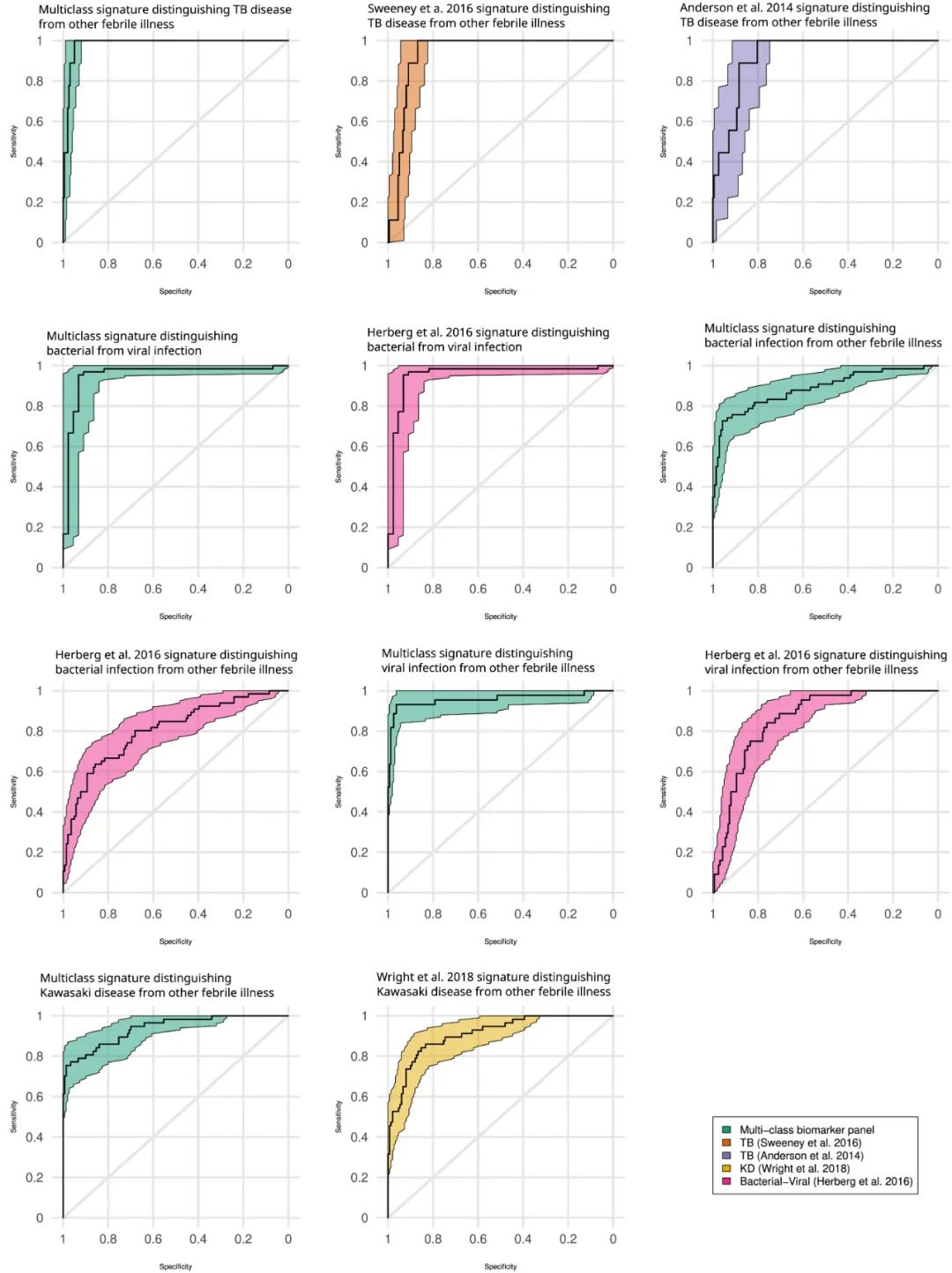
图5. 多RNA特征与已发布传染病特征的比较
结 语
该研究分析了是否可以使用单个全血转录组同时区分多种疾病。研究团队将多类机器学习方法应用于公开的血液基因表达数据集,以识别一组161个转录本,用于准确诊断儿童发热性疾病的多种原因。研究显示,161个转录组panel可以识别18种特定的炎症性疾病和病原体种类,并区分六大疾病类别(细菌感染、病毒感染、炎症性疾病、结核病、疟疾和川崎病),并成功地进行了从芯片发现队列到RNA-seq验证队列的跨平台和跨队列验证。
该研究提供了一个原则证明,即一组RNA转录本可以用于将发热和非特异性临床和实验室发现的患者归属于来自单一全血样品的一系列病因。结合诊断技术的进步,能够以可承受的成本快速检测RNA转录本。发热性疾病的多类生物标志物检测可以避免漫长的临床诊断过程,减少诊断延误、漏诊和不必要的抗生素治疗,可对全球健康产生重大影响。
研究人员强调,该方法尚无法用于临床实践,RNA转录组panel需要进一步调整、检测和验证并转化为易于使用的平台/设备,然后才能获得监管机构的批准。为了确保临床实用性,该方法的进一步发展将需要大量的前瞻性患者队列,具有一致、详细和准确的临床表型。通过扩大转录组发现中包括的病症范围,有可能改善大量患者的治疗,特别是对于罕见和诊断不足的病症。对于这些病症,早期检测和治疗可能具有显著的益处。类似地,考虑到适当的临床队列和基因表达数据集,有可能将这一原则扩展到其他人群,如成人、共病患者和受特定地理区域病原体(如登革热、虫媒病毒感染或莱姆病和斑疹伤寒等人畜共患疾病)影响的人群。
参考资料:
Habgood-Coote, D., Wilson, C., Shimizu, C., Barendregt, A. M., Philipsen, R., Galassini, R., ... & Kaforou, M. (2023). Diagnosis of childhood febrile illness using a multi-class blood RNA molecular signature. Med.
https://www.cell.com/med/fulltext/S2666-6340(23)00194-0?_returnURL=https%3A%2F%2Flinkinghub.elsevier.com%2Fretrieve%2Fpii%2FS2666634023001940%3Fshowall%3Dtrue


