NAR:伯晓晨课题组基于可解释机器学习模型,探索转录因子调控的发育和演化规律
时间:2024-06-11 06:02:17 热度:37.1℃ 作者:网络
作为生命的起点,受精卵蕴含着承载生命设计蓝图的遗传物质,在其发育分化为多细胞生物的过程中,遗传物质未发生改变,但发育形态却产生了巨大的变化。在物种的演化过程中,遗传物质指导基因表达进而控制物种间特异表现的性状,这一系列生命活动的背后是高度有序、精细的转录调控过程。转录因子(Transcription factor, TF)通过和顺式调控元件(Cis-regulatory elements, CREs)的相互作用来调控基因的时空表达,是转录调控过程的核心驱动因素。研究表明,胚胎发育和物种演化的大量差异表型不能仅用基因序列来解释,更多的是非编码区转录调控的结果。
在胚胎发育过程中,转录因子蛋白及其结合基序偏好性没有改变,但CREs的变化有助于调节发育可塑性。以往的研究发现转录因子在基因组中的分布并不均匀,转录因子结合位点(Transcription factor binding site,TFBSs)在人类、果蝇和线虫等真核生物的基因组中呈现高度聚集状态[1-6],揭示了基因组的区域化转录活性特征。然而,在跨物种的胚胎发育过程中,转录因子与CREs如何协同调控基因的时空表达仍然值得研究。
近日,军事医学研究院伯晓晨课题组在Nucleic Acids Research期刊上在线发表了文章“The developmental and evolutionary characteristics of transcription factor binding site clustered regions based on an explainable machine learning model”。

文章发表在Nucleic Acids Research
该研究收集了哺乳动物(人、小鼠、牛)、鱼类(斑马鱼和青鳉)和鸡处于胚胎发育阶段的染色质可及性数据和基因表达数据,对胚胎不同阶段的转录因子结合位点聚集区(Transcription factor binding site clustered regions,TFCRs)进行了识别,以研究其调控演进规律(图1)。
该工作通过跨物种比较,揭示了胚胎发育过程中TFCRs在物种间的分布规律:在胚胎发育过程中,TFCRs与启动子之间的距离变近;在进化过程中,从简单物种到复杂物种,TFCRs与基因之间的调控关系变得更加复杂。通过定义物种间的RegulatoryScore指数来评估TFCR复杂性和基因表达的协调一致性,发现人类胚胎干细胞中高调控得分的基因在神经元发育和突触传递过程中具有丰富的表达,表明与其他物种相比,人类发育过程中具有特定的神经元调控模式。
进一步,为了找出影响RegulatoryScore得分预测的因素,该研究使用了可解释的机器学习模型,发现在所有物种和发育阶段中,TFCRs与基因启动子之间的距离是影响TFCRs对基因表达调控的最大因素。综上,该工作揭示了从鱼类、鸡到哺乳动物胚胎发育过程中 TFCRs 的发育和进化动态,这些数据为探索胚胎发育过程中转录调控与表型差异之间的关系提供了资源,对于理解进化过程中胚胎发育阶段的转录调控具有重要的意义。
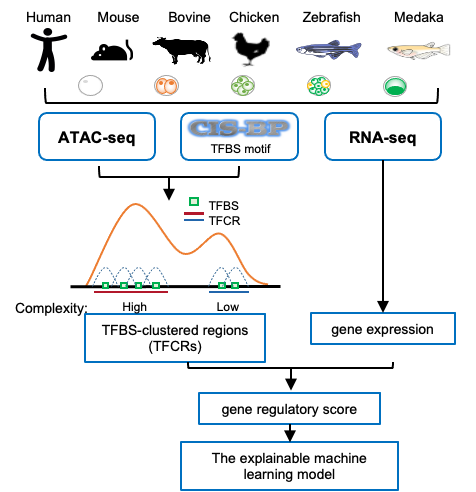
图1. TFCRs分析流程图
该研究首先描述了不同物种胚胎发育过程中TFCRs的性质。在比较TFCR时,不同的测序深度可能会导致偏差。通过系统评估测序深度对识别结果的影响,研究团队证明了在不同测序深度下,TFCR的识别是可靠的(图2)。
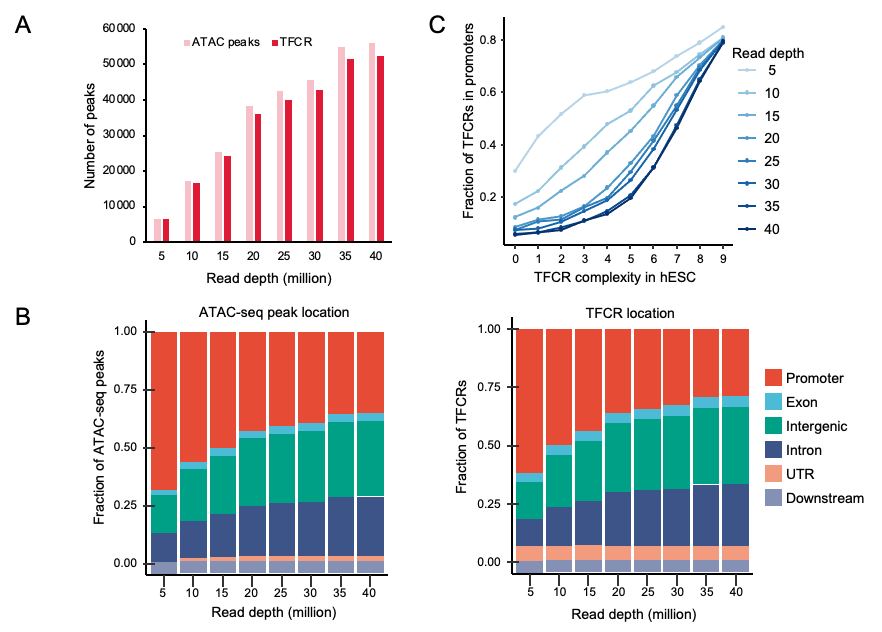
图2. 不同测序深度数据中TFCR的识别稳定性
超级增强子(Super-enhancers,SEs)是基因组中一个极度活跃的CREs。增强子和SE在远端调节基因的表达,这可以决定细胞的特性和谱系特异性。该研究通过比较hESC中的TFCRs和SEs,发现SEs的数量低于TFCRs,而SEs的长度大于TFCRs。在hESC中,56%的增强子与TFCRs重合,31%的SEs位于最复杂的TFCR9。这种高重合率的一个可能解释是TFCRs在SEs中起着关键作用。由于其长度较短,TFCRs在定位功能序列方面可能比SEs更准确。该研究还发现SEs和TFCR9都富含管家基因和癌症相关基因;但SE的癌基因比例高于TFCR9,TFCR9的管家基因比例高于SE。此外,SE丰富了必需基因,而TFCR9则丰富了相对较老的基因。
通过比较TFCRs在基因组分布(图3),研究团队发现相比于鸡和鱼类(斑马鱼、青鳉),人类的TFCRs在启动子区域更富集,这表明人类的转录调控模式可能更倾向于近端和复杂的转录调控;鸡和鱼TFCRs在基因间区的富集表明其可能倾向于通过增强子进行远端调控。人类中更复杂的TFCRs为更多的TF参与到基因调控的过程提供了先天条件,表明其调控模式比鸡和鱼类更复杂。当一个转录因子不能与其结合时,类似的转录因子仍然可以启动基因转录,显示出转录调控的鲁棒性。
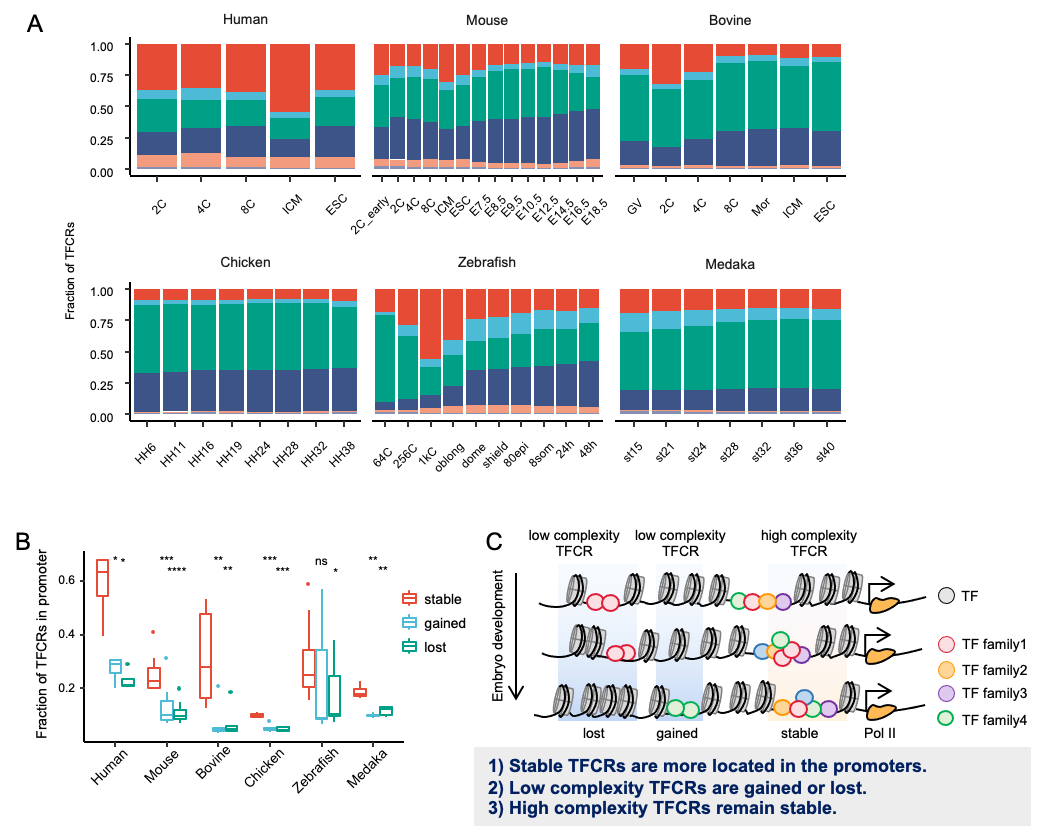
图3. 不同物种中TFCRs的分布特性
该研究在不同物种均发现受精卵基因激活期的转录调控变化剧烈而胚胎发育后期相对稳定。稳定的TFCRs在胚胎发育过程中具有更高的复杂性,可能有更复杂的调控来维持一些必要的生物学过程。相比之下,动态TFCR的复杂性较低,可能受某些特定转录因子的调控,这些转录因子仅在特定的阶段参与;并且胚胎发育不同阶段间稳定的TFCRs具有更高的复杂性,比动态变化的TFCRs位于启动子的比例更高(图3)。
从简单物种到复杂物种,不仅CREs的序列发生了很大的变化,TFs和CREs的调控模式也发生了变化。为了衡量物种之间调控模式的差异,该研究定义了RegulatoryScore衡量TFCRs复杂度和基因表达量的一致性,并通过基因功能富集分析发现,RegulatoryScore较高的基因能够体现出其相应阶段的生物过程。
为了探索序列特征和RegulatoryScore之间的隐藏关系和模式,该研究使用了11种不同的基于机器学习的方法,包括CatBoost、支持向量回归(SVR)、卷积神经网络(CNN)等,基于启动子序列、TFCR序列和它们之间的距离来预测RegulatoryScore;最后基于预测效果较好的CatBoost模型,使用了SHAP分析以解释所有输入特征对于CatBoost模型预测的重要性。
结果表明,TFCRs与启动子之间的距离是影响TFCRs调控基因在物种和发育阶段间表达的最重要因素,预测的RegulatoryScore随着距离的增加而减少,并且在所有物种和所有阶段之间存在显著的负相关。上述研究结果揭示了鱼类、鸡到哺乳动物胚胎发育过程中TFCRs的发育和进化动态,从可解释机器学习模型的角度阐明了TFCRs与基因启动子的距离对于基因调控的重要性,这些数据为探索胚胎发育过程中转录调控与表型差异之间的关系提供了资源。
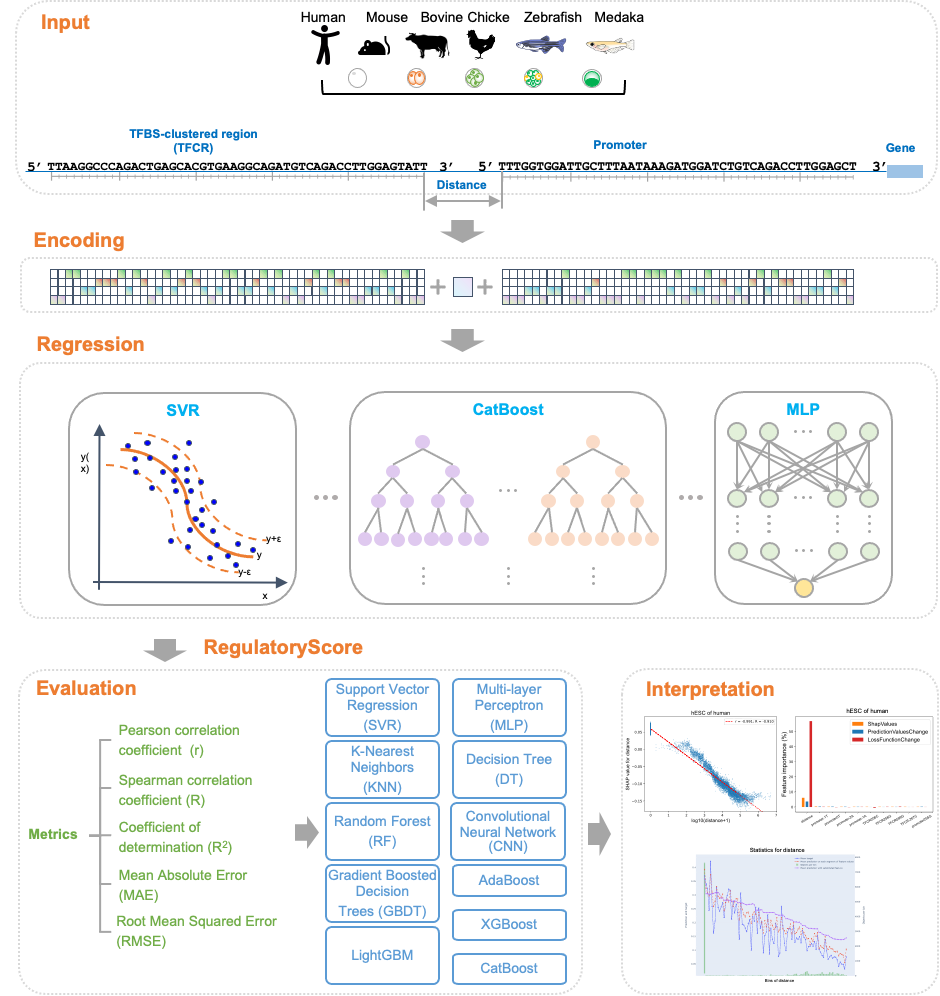
图4. 可解释机器学习分析RegulatoryScore特征重要性
基因组构象是影响转录调控的关键因素。先前的研究发现一些TFCRs在空间上是相邻的[7],这表明TFCRs可能与基因组三维结构的变化相关。近来多篇研究报告了三维基因组构象在进化过程中调节基因表达的功能[8,9]。随着未来更多物种胚胎Hi-C数据的积累,TFCRs在跨物种早期胚胎中的转录调控机制及其在基因组三维构象的作用有待进行更详细的研究。
军事医学研究院陈河兵副研究员、伯晓晨研究员、李昊副研究员和从玉文研究员为论文的共同通讯作者,助理研究员欧阳张翼、重庆医科大学刘峰副研究员、硕士研究生李宛莹为并列第一作者。北京大学李程研究员为该工作提供了重要指导。研究生王军婷、陈碧嘉、郑炀、李雅儒、陶欢、许翔参与了这项工作。
Reference:
1.Iwasaki, Y., Wada, K., Wada, Y., Abe, T. and Ikemura, T. (2013) Notable clustering of transcription-factor-binding motifs in human pericentric regions and its biological significance. Chromosome Res, 21, 461-474.
2.Yan, J., Enge, M., Whitington, T., Dave, K., Liu, J., Sur, I., Schmierer, B., Jolma, A., Kivioja, T., Taipale, M. et al. (2013) Transcription factor binding in human cells occurs in dense clusters formed around cohesin anchor sites. Cell, 154, 801-813.
3.Negre, N., Brown, C.D., Ma, L., Bristow, C.A., Miller, S.W., Wagner, U., Kheradpour, P., Eaton, M.L., Loriaux, P., Sealfon, R. et al. (2011) A cis-regulatory map of the Drosophila genome. Nature, 471, 527-531.
4.Niu, W., Lu, Z.J., Zhong, M., Sarov, M., Murray, J.I., Brdlik, C.M., Janette, J., Chen, C., Alves, P., Preston, E. et al. (2011) Diverse transcription factor binding features revealed by genome-wide ChIP-seq in C. elegans. Genome Res, 21, 245-254.
5.Chen, H., Li, H., Liu, F., Zheng, X., Wang, S., Bo, X. and Shu, W. (2015) An integrative analysis of TFBS-clustered regions reveals new transcriptional regulation models on the accessible chromatin landscape. Sci Rep, 5, 8465.
6.Tang, X., Wang, J., Tao, H., Yuan, L., Du, G., Ding, Y., Xu, K., Bai, X., Li, Y., Sun, Y. et al.(2022) Regulatory patterns analysis of transcription factor binding site clustered regions and identification of key genes in endometrial cancer. Comput Struct Biotechnol J, 20, 812-823.
7.Chen, H., Jiang, S., Zhang, Z., Li, H., Lu, Y. and Bo, X. (2017) Exploring spatially adjacent TFBS-clustered regions with Hi-C data. Bioinformatics, 33, 2611-2614.
8.Alvarez-Gonzalez, L., Arias-Sarda, C., Montes-Espuna, L., Marin-Gual, L., Vara, C., Lister, N.C., Cuartero, Y., Garcia, F., Deakin, J., Renfree, M.B. et al. (2022) Principles of 3D chromosome folding and evolutionary genome reshuffling in mammals. Cell Rep, 41, 111839.
9. Li, D., He, M., Tang, Q., Tian, S., Zhang, J., Li, Y., Wang, D., Jin, L., Ning, C., Zhu, W. et al. (2022) Comparative 3D genome architecture in vertebrates. BMC Biol, 20, 99.


