Nature子刊:戴琼海/邓岳团队构建多模态融合的深度生成模型soScope,可高分辨率表征复杂组织结构
时间:2024-08-18 06:02:31 热度:37.1℃ 作者:网络
导读
近年来,空间组学技术的重大进步使得各种分子的空间分析成为可能,包括转录本(Visium、seqFISH等)、蛋白质(spatial-CITE-seq)、表观遗传标记(spatial ATAC-seq、spatial-CUT&Tag等)和基因组变异(slide-DNA-seq和 DNA-seqFISH),为不同生物学领域提供了宝贵见解。
但仍有两个挑战限制了空间组学技术的应用:一是组织样本经冷冻或福尔马林固定、石蜡包埋(FFPE)处理可能会影响分子状态,从而导致测序准确性降低;二是大多数空间技术在组织点分辨率下进行分析,忽略了异质性,导致组织结构的空间分辨率有限。目前开发的可提高空间分辨率的计算方法主要针对转录组学数据,缺乏与其他新兴空间组学技术的适应性。
近日,清华大学成像与智能技术实验室戴琼海院士团队与北京航空航天大学人工智能学院邓岳教授团队合作在Nature Communications上发表了题为“Tissue characterization at an enhanced resolution across spatial omics platforms with deep generative model”的文章。研究团队开发了一个统一的生成式模型“soScope”,可提高从各种空间技术获得的分子图谱数据质量和空间分辨率。soScope可整合来自组学、空间关系与图像的多模态组织信息,并通过分布先验与组学特异性建模共同推断出分辨率增强的组学图谱。
通过将soScope与多种空间组学平台进行综合评估,发现soScope提高了识别具有生物学意义的肠道和肾脏结构的性能,揭示了无法原始分辨率下无法解析的胚胎心脏结构,并纠正了因测序和样本处理而产生的样本及技术偏差。此外,soScope还可扩展到空间多组学技术spatial-CITE-seq和spatial ATAC-RNA-seq中,利用跨组学参考同时进行多组学增强。综上,soScope是一种多功能工具,可提高对不断扩展的空间组学技术和资源的利用率。

文章发表在Nature Communications
主要内容
1 soScope技术介绍
soScope是一个完全生成式模型(图1),可对来自不同空间组学技术的点级剖面(profile)生成过程进行建模,提高其空间分辨率和数据质量。soScope将每个spot(点)视为空间分辨率增强的“subspots(子点)”的集合,其组学特征与空间位置、形态模式相关联;随后利用多模态深度学习框架将spot组学剖面、空间关系和高分辨率形态图像进行整合,并在子点分辨率下联合推断组学图谱。通过选择组学特异性分布,soScope可对不同空间组学数据进行精确建模并减少变异。
soScope提供了一个统一的工具,可整合多模态组织图谱,来增强不同分子类别的组学图谱。通过对来自多个生物系统的不同空间组学类型(转录本、表观遗传学、DNA 和蛋白质)进行综合基准测试,研究团队证明soScope可有效地生成组学图谱,同时降低数据噪声并增强空间分辨率,从而更精细地表征组织结构。此外,soScope还可扩展到新兴的空间多组学技术,整合多模态、多组学组织图谱并同时提高其分辨率。
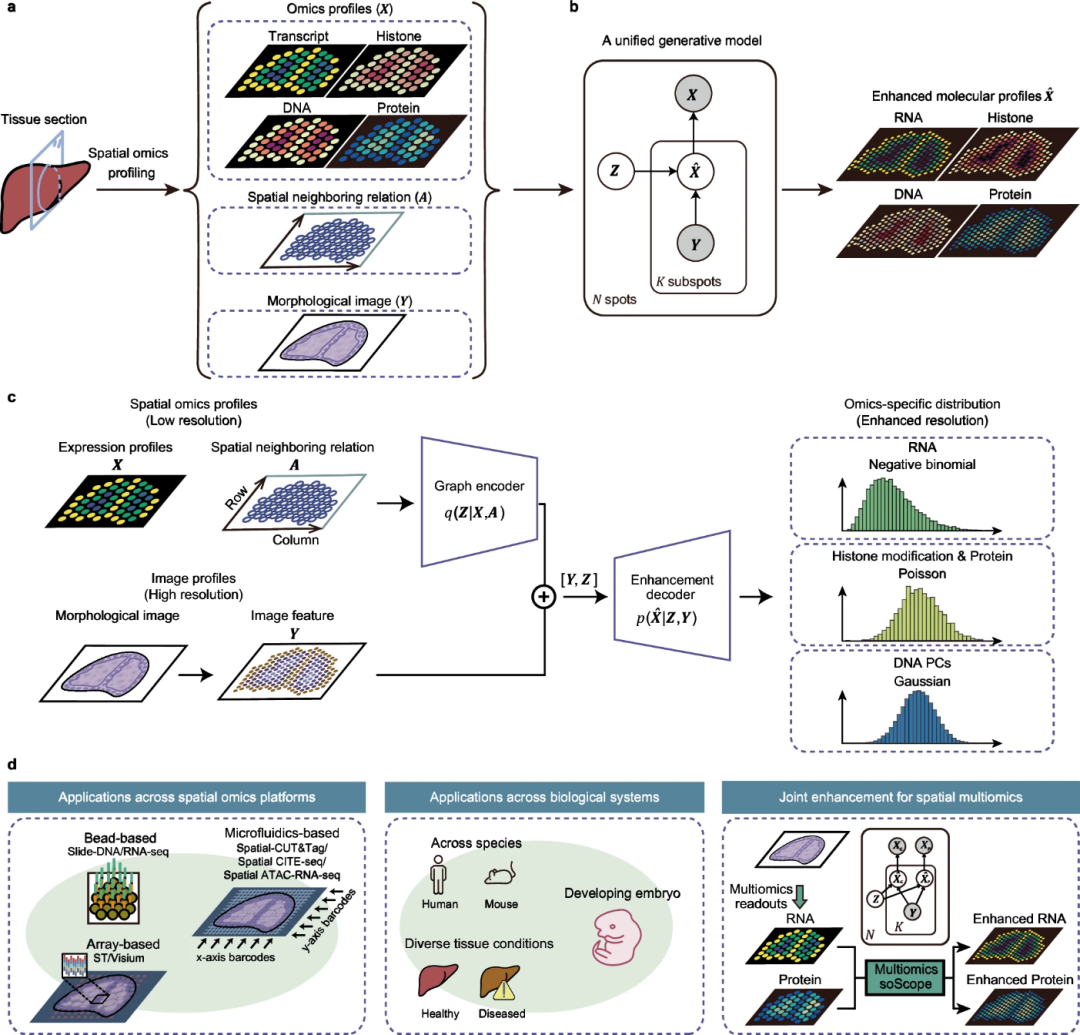
图1.soScope及其应用概述
2 soScope优于为空间转录组学开发的分辨率增强方法
研究团队将soScope与三种空间组学分辨率增强方法进行了比较(图2a),即iStar、XFuse和BayesSpace,使用一个仅使用图像输入的soScope(image soScope)作为参考;通过合并来自邻近点的转录数据来模拟“低分辨率”表达图谱,并通过比较重建的表达和原始图谱来评估分辨率增强方法的性能。
研究团队首先使用Visium平台生成的人类肠道数据集对上述方法进行评估(图2b-2e),其包含2,649个测序点;依据基准设计将这些测序点合并成369个“低分辨率”点;并重点检查了肠道组织中上皮、肌层和免疫等三个功能区域。结果显示,iStar在免疫区域表现良好,但未能反映上皮和肌层的精细结构;XFuse在肌层区域表现良好,但未能完整保留上皮和免疫区域的组织结构;BayesSpace无法细化剖面区域边界。
相比之下,soScope在三个区域都取得了不俗的表现,不仅完整重建了人类肠道组织的精细结构,还始终表现出最高的一致性和最小的重建误差。进一步分析显示,soScope和iStar在大多数测试基因中产生了最明显的分离效果,并实现了与原始图谱相似的可区分性。
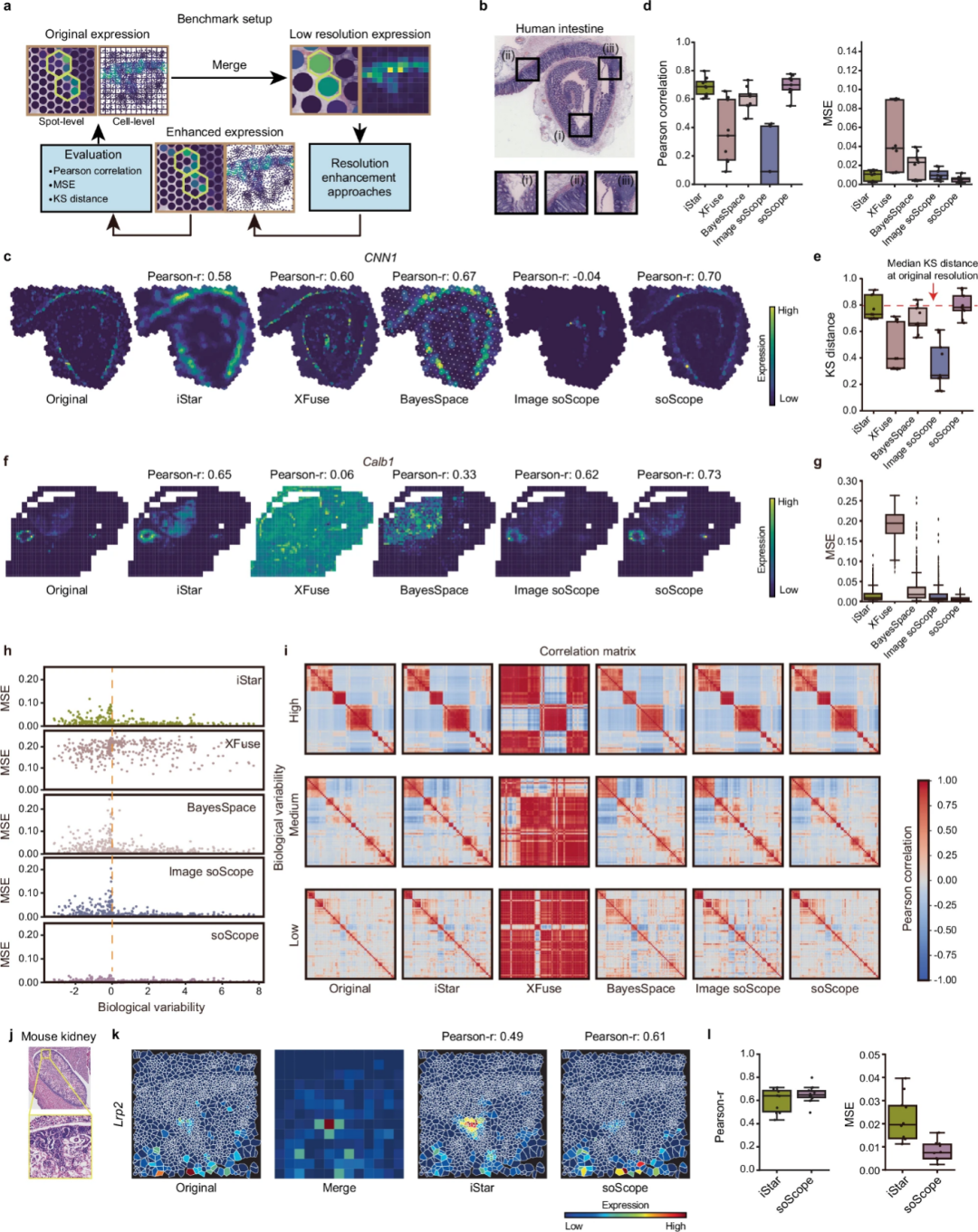
图2.在来自多个组织和平台的空间转录组学数据集上评估soScope
接下来,研究团队使用从Xenium平台获得的小鼠数据集进行性能评估(图2f-2i),共纳入379个测试基因;将来自原始细胞级转录本的2,619个点合并为291个“低分辨率”点,用于分辨率增强分析。结果显示,iStar和soScope在保留高分辨率表达谱方面均表现出色,但soScope在不同基因变异中显示出更稳定的性能。除XFuse之外,大多数方法在分辨率增强后仍能保留基因间的相关性模式,而不受生物学变异性差异影响,其中soScope的相关性最高。
研究团队还使用了Xenium平台的小鼠肾脏数据集,评估了iStar、soScope增强单细胞分辨率的能力(图2j-2l)。结果显示,iStar倾向于高估高细胞密度和高表达区域的表达水平,soScope显示出与真实情况更一致的表达模式。此外,大多数方法的精度会随着分辨率的降低而降低,但soScope在不同分辨率下始终表现出稳定的性能。
上述结果表明,soScope有能力跨多空间技术平台、分辨率要求和基因统计特征提高转录数据的空间分辨率,从而实现对组织结构的详细表征。
3 soScope生成小鼠胚胎详细结构
除传统转录组学外,通过将生成过程与组学特定分布相结合,soScope可灵活地扩展到不同组学类型中。为验证这种多功能性,研究团队将soScope应用于由spatial-CUT&Tag生成的空间染色质可及性数据集中,这些数据从小鼠胚胎上收集,包括1974个测序点;从四个主要器官区域(肝脏、心脏、前脑和脊髓)中选择前60个可变峰进行分辨率增强分析,并与六种通用机器学习模型进行比较(图3)。
在保持原始分辨率下,soScope在峰值计数方面取得了最高的相关性和最低的重建误差。当原始数据的组织分辨率提高4倍时,其他方法均受到噪声导致性能大幅下降,但soScope同时结合了相邻点之间的空间关系和图像参考,在有效降低测序噪声的同时准确正确识别了表达区域。
接下来,研究团队使用soScope对心脏区域进行重点研究,其可进一步分为小梁心室心肌(标志基因Fhl2)和致密心室心肌(标志基因Ldha),但在低分辨率下无法观察到。结果显示,soScope的增强峰值分布反映了心脏的两层结构:Fhl2在小梁心室心肌中表现出高活性,Ldha在致密心室心肌中表现出高活性。上述结果显示,soScope的生成建模提高了数据质量,并可捕获精细的组织结构。
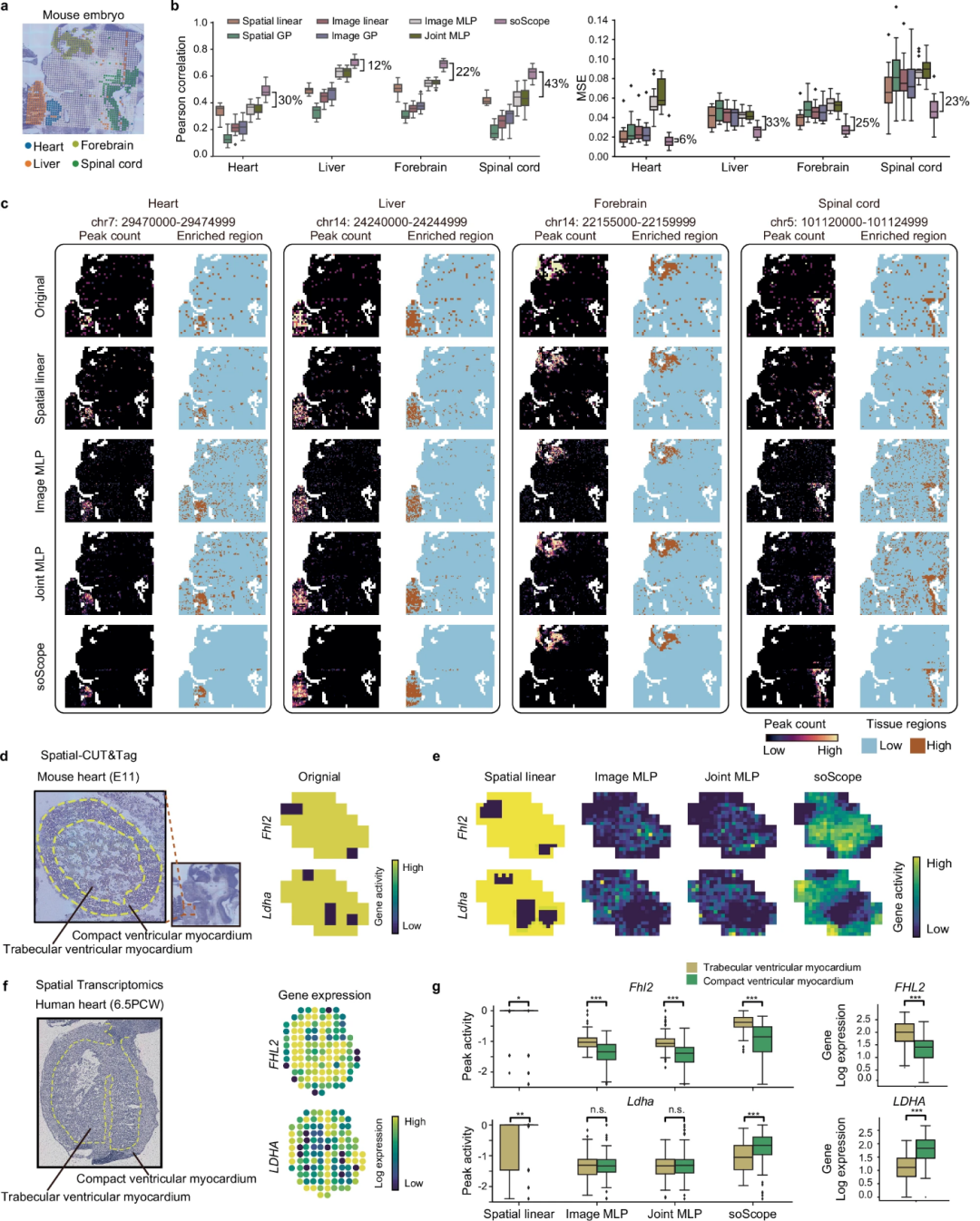
图3.在小鼠胚胎的空间染色质可及性数据集上评估soScope
4 多组学soScope利用跨组学参考纠正技术偏差
空间多组学的最新进展拓展了人们在单一组学类型之外进行空间组织分析的能力,现有开创性的技术包括spatial-CITE-seq和spatial ATAC-RNA-seq。同时收集多个分子特征有可能克服单一组学的局限性,并揭示更完整的组织结构。
为此,研究团队对soScope进行了改进(称为multi-soScope),使其能够处理空间多组学,并将其分别应用于从人体皮肤组织中收集的spatial-CITE-seq数据集和从小鼠胚胎获得的spatial ATAC-RNA-seq数据集中(图4)。结果显示,通过使用不同空间组学平台生成的不同分子类别数据集,multi-soScope能够有效地整合多组学图谱,补偿质量较低的组学数据,并联合提高多组学的分辨率。上述结果表明,soScope可用于空间多组学的联合分析中,并实现不同数据类型间的协同优化。
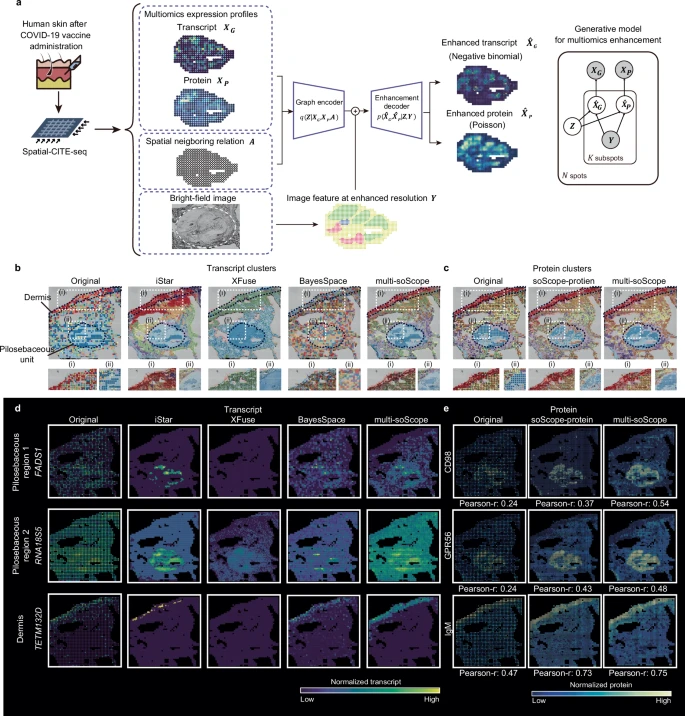
图4. multi-soScope在人体皮肤组织的空间蛋白+转录本数据集中的应用
综上所述,该研究开发出一个强大的组学分辨率增强工具——soScope,可结合组学图谱、空间邻近关系和来自同一组织的形态学图像,通过对组学特有的数据变化建模,以更高的分辨率推断组学图谱。研究团队广泛评估了soScope在多种空间技术上的有效性和通用性,发现其细化了组织结构域识别,提高了已知标记的可区分性,并纠正了数据和技术偏差。随着可用空间组学数据资源的不断扩大和新空间技术的出现,soScope可作为一个多功能工具,充分利用空间组学数据,增强人们对复杂组织结构和生物过程的理解。
论文原文:
Li, B., Bao, F., Hou, Y. et al. Tissue characterization at an enhanced resolution across spatial omics platforms with deep generative model. Nat Commun 15, 6541 (2024).
https://www.nature.com/articles/s41467-024-50837-5


