Nature Microbiology:上海交通大学医学院公共卫生学院院长王慧联合多团队发现结直肠癌诊断的四界微生物组合标志物
时间:2022-02-02 10:18:14 热度:37.1℃ 作者:网络

近日,上海交通大学医学院公共卫生学院王慧教授联合同济大学朱瑞新教授、中科院上海营养与健康研究所张国庆教授、复旦大学陈兴栋教授和中山大学附属第六医院朱立新教授团队,在国际知名期刊《自然微生物》杂志(Nature Microbiology)上发表了题为Multi-kingdom microbiota analyses identify bacterial–fungal interactions and biomarkers of colorectal cancer across cohorts的研究论文,该研究针对结直肠癌患者肠道菌群的四界微生物组进行多中心队列研究,分析和绘制了全球八个结直肠癌人群队列的四界微生物组图谱,发现结直肠癌特异的细菌-真菌互作新机制,为结直肠癌诊断提供了基于微生物及其功能标志物的新方法。
癌症是全球主要的公共卫生问题[1],且成为我国第三大、美国第二大死亡原因。《临床肿瘤杂志》“CA”期刊发布的2021年癌症统计报告显示,无论是在男性还是女性中,结直肠癌的发病率和死亡率都位居第三,极大地加重了社会负担,因此亟需新的结直肠癌早筛方法和治疗方案用于临床[2-3]。最近大量研究表明,结直肠癌的发生发展与肠道微生物组的改变密切相关[4-5],而肠道微生物组包括细菌、真菌、古菌和病毒四界微生物组,因此,对于结直肠癌发生发展的机制围绕肠道微生物组四个界别的微生物开展整体的、客观的和系统的研究,尚属空白。
该研究整合了全球8个国家/地区的结直肠癌队列共1368例标本的宏基因组数据,不仅发现了可用于结直肠癌诊断的单界别微生物菌株标志物(27个细菌、20个真菌、20个古菌和21个病毒物种),还首次发现了准确性更高的多界别最小核心菌群(11个细菌、4个真菌和1个古菌),此外,通过宏基因组功能预测,结直肠癌患者肠道菌群的D-氨基酸代谢和丁酸盐代谢水平显著升高。总之,该研究不仅揭示了不同地域人群中普遍存在的结直肠癌特异的微生物标志物,同时验证了四界微生物组作为结直肠癌诊断辅助工具的可行性,并进一步提出了微生物及其相关功能基因作为治疗结直肠癌潜在靶点的可能性,为结直肠癌早期诊断和预后评估提供了基于微生物及其功能标志物的新方法和新思路。
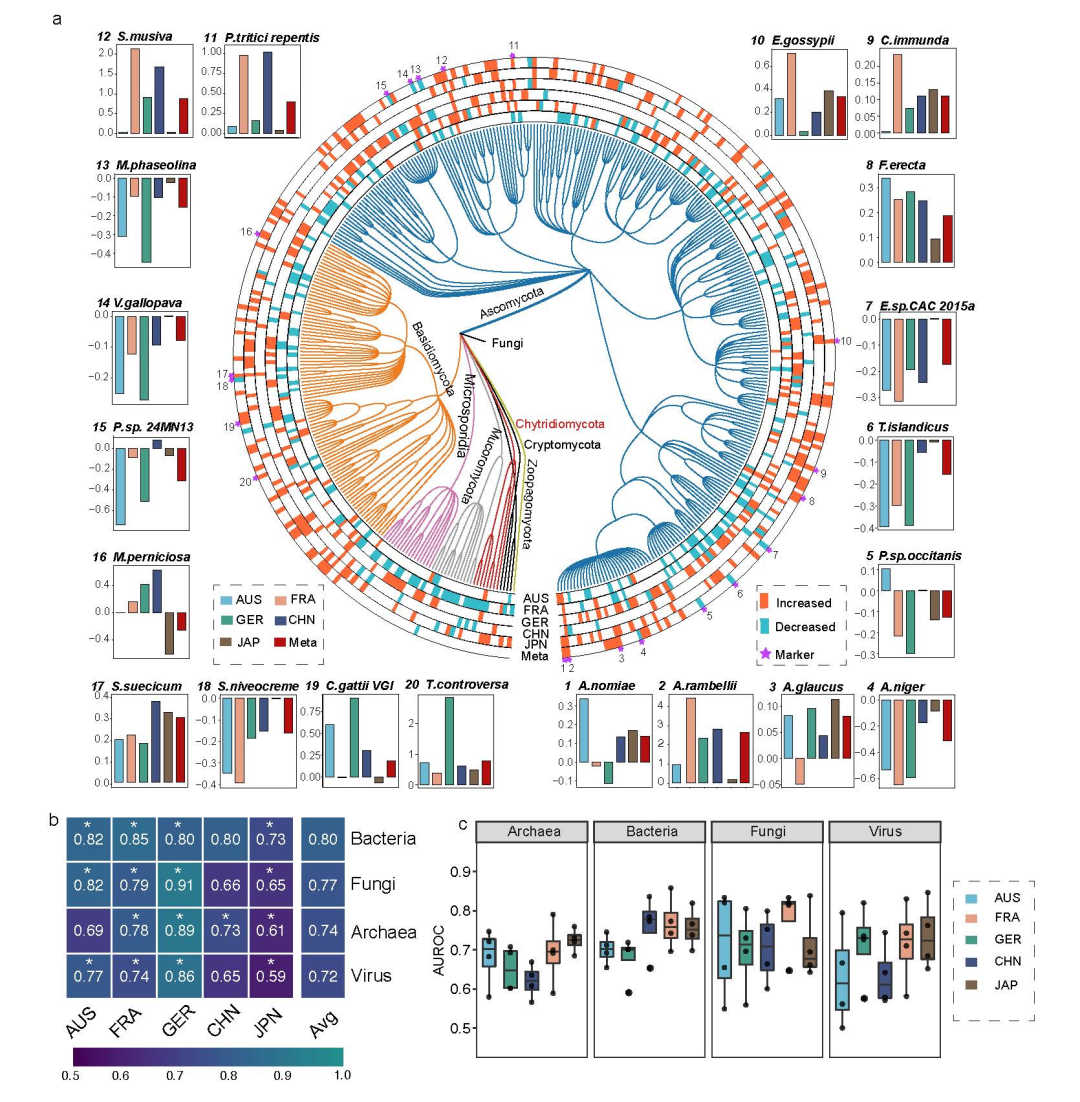
图1. 多中心队列中共有的结直肠癌特异性真菌标志物及其诊断能力预测
该研究得到了国家科技部重点研发计划、国家自然科学基金、上海市科委和卫健委等项目的资助,第一作者和最后通讯作者单位为上海交通大学医学院。上海交通大学医学院公共卫生学院刘宁宁研究员、浙江大学医学院附属儿童医院焦娜研究员、上海交通大学医学院公共卫生学院硕士研究生谭镜璁、上海市中医医院王子良研究员为论文的共同第一作者。该项目还得到了合作方中国科学院赵国屏院士、海军军医大学廖万清院士、海军军医大学潘炜华教授、海军军医大学方文捷博士、美国农业部农业研究局Geromy G. Moore教授、上海交通大学医学院单细胞组学与疾病研究中心Christopher Heeschen教授、世界卫生组织国际癌症研究署Zisis Kozlakidis教授的大力支持和帮助。
感谢论文三位审稿人 Ran Blekhman, Aleksandar Kostic 和George Miller,对该项工作给予了高度评价:“All my concerns have been addressed. I believe that this is an important contribution.” “I would like to congratulate the authors on this interesting and comprehensive study. My concerns have been sufficiently addressed.”
原文链接:
https://www.nature.com/articles/s41564-021-01030-7
参考文献
1. Siegel RL, Miller KD, Fuchs HE, Jemal A. Cancer Statistics, 2021. CA Cancer J Clin. 2021 Jan;71(1):7-33. doi: 10.3322/caac.21654. Epub 2021 Jan 12. Erratum in: CA Cancer J Clin. 2021 Jul;71(4):359.
2. Dekker, E., Tanis, P. J., Vleugels, J. L. A., Kasi, P. M. & Wallace, M. B. Colorectal cancer. Lancet 394, 1467–1480 (2019).
3. Keum, N. & Giovannucci, E. Global burden of colorectal cancer: emerging trends, risk factors and prevention strategies. Nat. Rev. Gastroenterol. Hepatol. 16, 713–732 (2019).
4. Janney, A., Powrie, F. & Mann, E. H. Host–microbiota maladaptation in colorectal cancer. Nature 585, 509–517 (2020).
5. Yu, T. et al. Fusobacterium nucleatum promotes chemoresistance to colorectal cancer by modulating autophagy. Cell 170, 548–563.e16 (2017).


